hifiasm 是一种快速的单倍型解析从头组装工具,最初专为 PacBio HiFi 测序数据设计。其最新版本支持利用超长 Oxford Nanopore 测序数据进行端粒到端粒(telomere-to-telomere, T2T)组装。Hifiasm 结合 HiFi、超长测序数据和 Hi-C 数据,能够生成目前最优质的单样本端粒到端粒组装结果。此外,在提供父本和母本的 NGS 数据的情况下,它也是分型组装(trio-binning)中最优秀的单倍型解析组装工具之一。对于人类基因组,Hifiasm 可在一天内完成端粒到端粒组装。
目前 hifiasm 的 latest 版本号为 0.24.0-r702, 从 0.20.0-r639 版本以来,hifiasm 做了不少有关纠错部分及 ONT 组装部分的大更新,同时作者表示,高深度 (>60 X) 数据产生次优组装结果是已知问题,并提供了临时解决方案。由于我之前使用的版本是 0.19.9-r616, 对于这几个版本的更新效果非常好奇,于是准备了高深度数据对比组装结果,尤其以 0.19.9, 0.24.0 两个版本的比较为主。
本次测试使用数据为 SRR26555721, 来自西瓜泛基因组的文章;HiFi base 约为 54.1G, 文章中组装大小为 364.5 M, 数据深度约 148 X.
组装结果
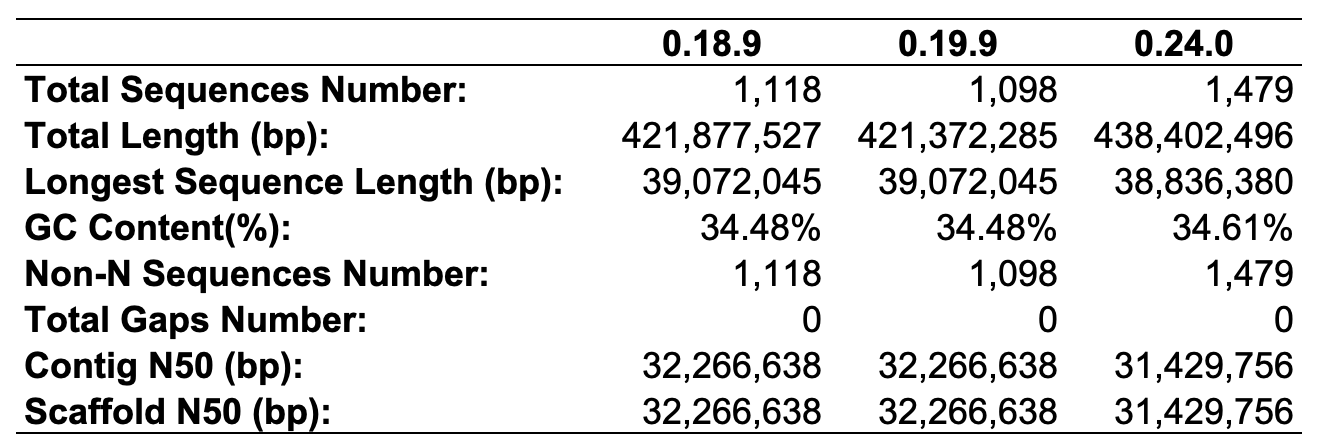
组装结果统计
首先对组装结果做个基本统计:
这里用相同的数据跑了三个版本的 hifiasm, 全部使用默认参数,不指定 -l 或 -n 等。可以看到 0.18.9 版本和 0.19.9 版本组装结果基本一致,N 50 水平一致,总 contig 数量也差不多。而 0.24.0 版本的结果相比之下反而差一些,contig 数量较多,N50 反而会低一点。
然后看一下 N50-N90 水平,0.24.0 版本的结果也没什么优势。
三代数据回比
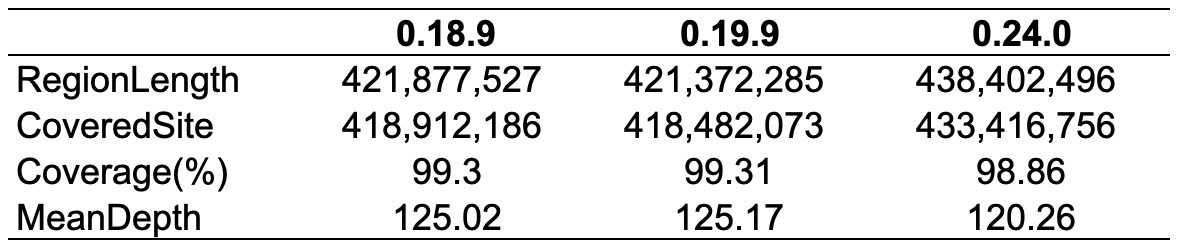
为了进一步比较组装质量,我将三代数据回比到了组装结果上,minimap2 比对参数为 -x map-hifi -x asm20 --MD -a 没什么特殊的,随后使用 samtools 获得 .sort.bam , 并使用 pandepth 统计覆盖度深度,有意思的来了:
98.8%本身很不错了,就怕别人都是 99+; 这个原因我们暂且按下不表,先看看别的评估结果;
端粒信号鉴定
使用 quarTeT 鉴定端粒特征信号,特征序列使用 AAACCCT, 统计结果如下:
西瓜本身 11 条染色体,总计 22 个端粒,不同版本总计获得 20 个左右端粒信号,整体比较合理。
问题排查
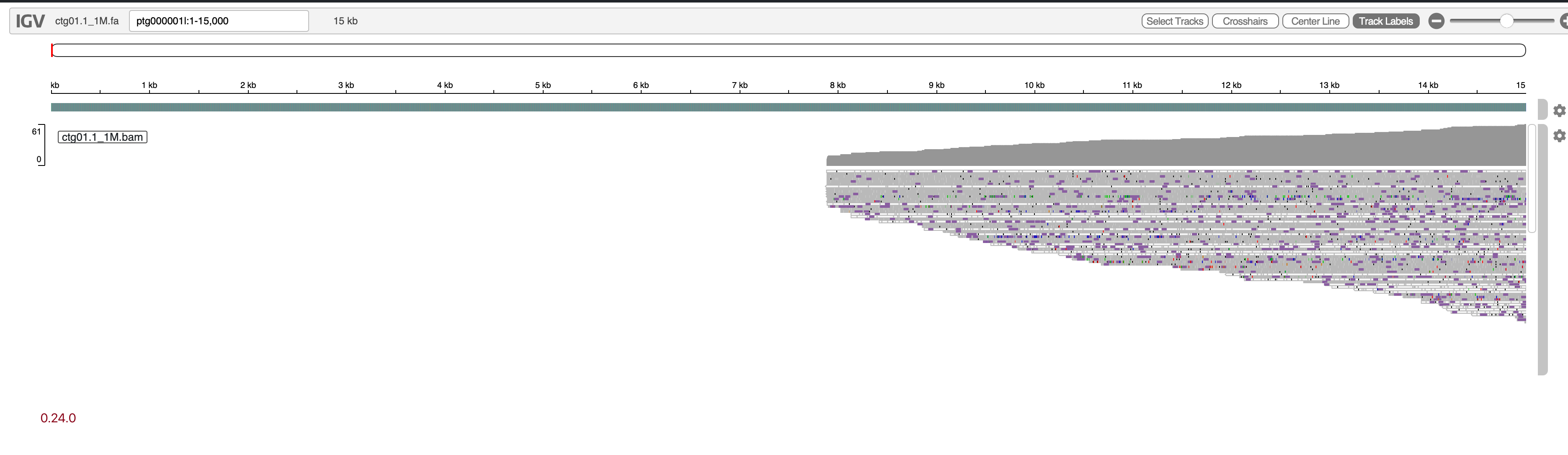
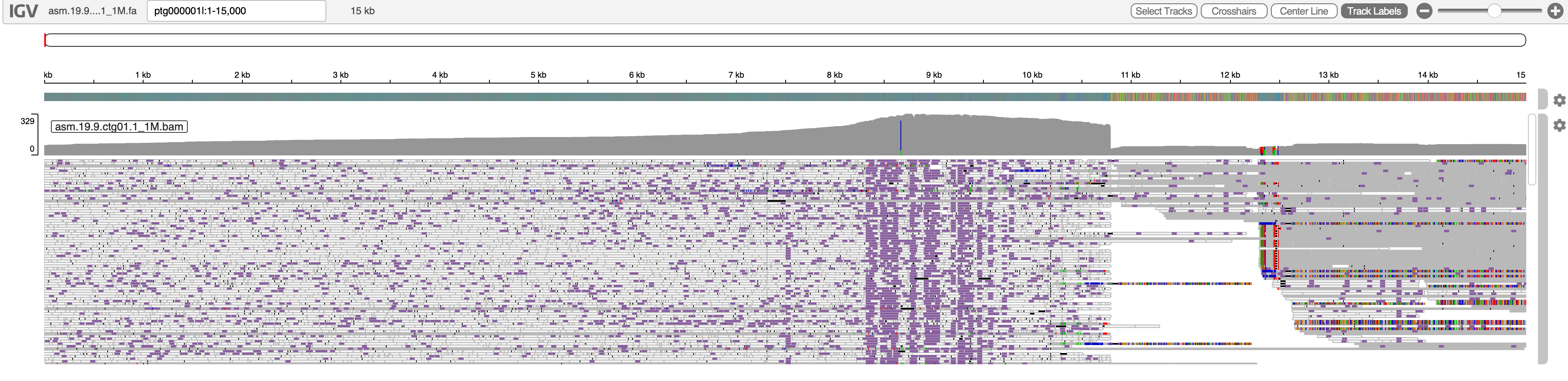
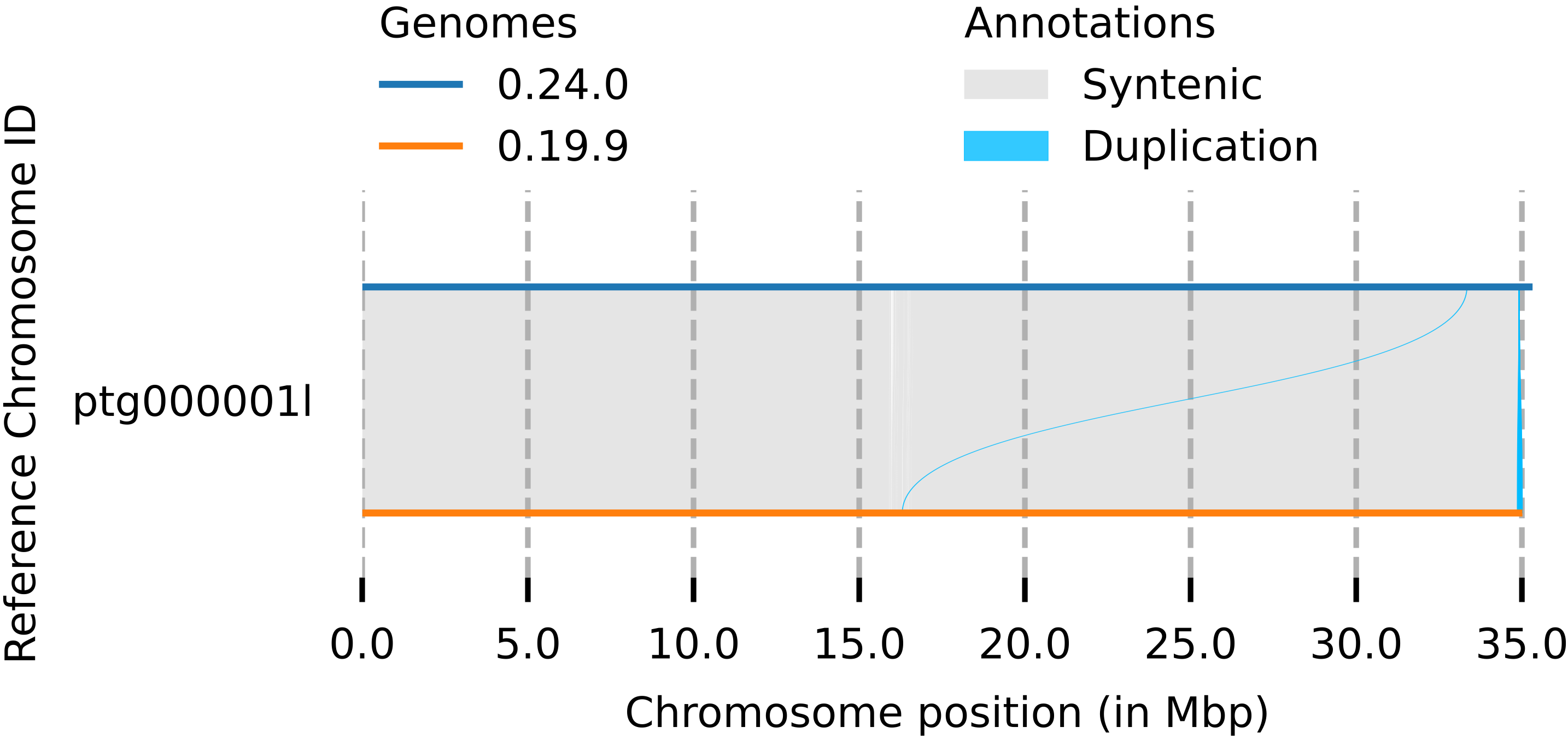
首先我好奇为啥 0.24.0 组装版本三代数据回比结果会低一些?查看了具体比对结果后,发现部分回比结果异常,这里主要比较 0.19.9 版本的结果,与 0.24.0 版本的结果。以 ptg000001l 为例:
这里上图是 0.24.0 版本的结果,下图是 0.19.9 版本的结果
可以看到,0.24.0 版本的组装结果存在一些无 reads 覆盖的区域,对于我这个例子甚至集中在端粒区。对于本身组装结果,两个版本的组装大小如下:
- v0.24.0: 35,325,984 bp
- v0.19.9: 35,015,304 bp
而上面我使用 quarTeT 鉴定了端粒重复序列,对于两个 ptg000001l, 起始端重复单元重复次数如下:
- v0.24.0: 2,684 times
- v0.19.9: 1,529 times
可以看到,0.24.0 版本的结果相比 0.19.9 版本的结果大了 300 kb 左右,但是差异又不全部来自端粒序列,这里我对两条 contig 用 mumer4 做了比对,结果如下:
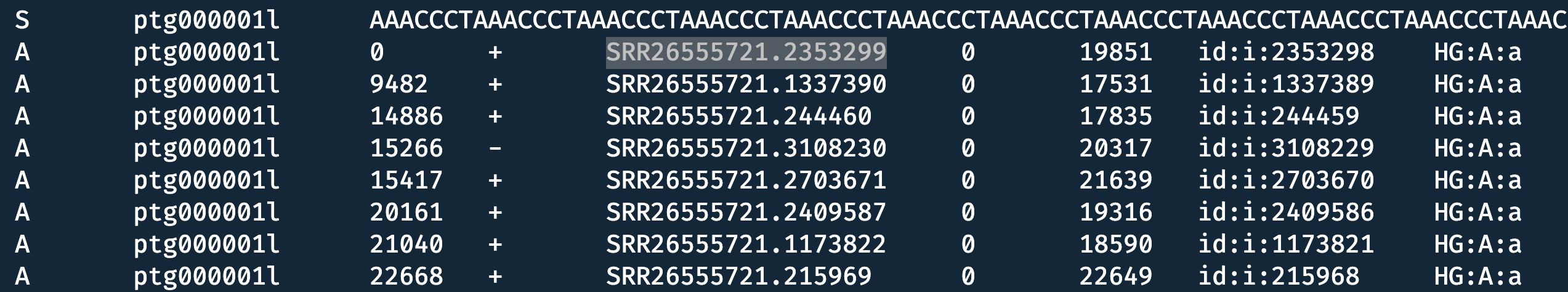
对于这个问题我在 hifiasm 的 GitHub 主页上提了 issue, 作者建议我看一下 gfa 中的结果,这里可以看到,对 0.24.0 版本的结果,用于组装的 reads 没有比对回对应的位置:
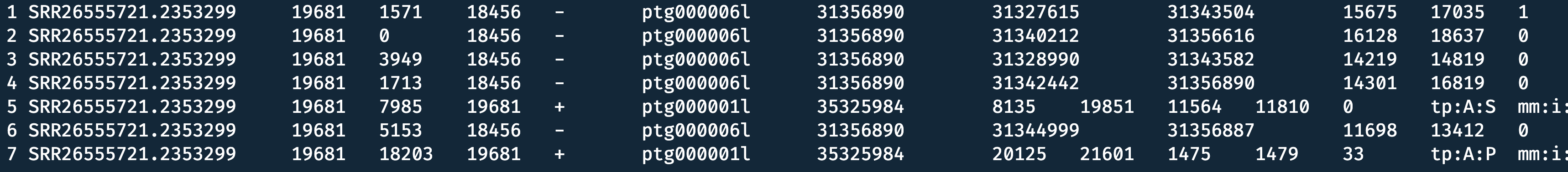
比对 paf 文件中,显示这个 reads 比对到了其他地方:
目前这个问题没有其他进展,很难讲会不会是比对的问题;我自己的话大概会继续使用 0.19.9 版本,等待这个问题有进一步答复。issue 的链接放在这里,有进展我也会及时同步。